Free Statistics
of Irreproducible Research!
Description of Statistical Computation | |||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Author's title | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Author | *The author of this computation has been verified* | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| R Software Module | rwasp_edauni.wasp | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Title produced by software | Univariate Explorative Data Analysis | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Date of computation | Sun, 26 Oct 2008 08:53:24 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cite this page as follows | Statistical Computations at FreeStatistics.org, Office for Research Development and Education, URL https://freestatistics.org/blog/index.php?v=date/2008/Oct/26/t1225033035p1kql5znwvckul6.htm/, Retrieved Sat, 18 May 2024 19:52:30 +0000 | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Statistical Computations at FreeStatistics.org, Office for Research Development and Education, URL https://freestatistics.org/blog/index.php?pk=18910, Retrieved Sat, 18 May 2024 19:52:30 +0000 | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| QR Codes: | |||||||||||||||||||||||||||||||||||||||||||||||||||||
|
| |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Original text written by user: | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| IsPrivate? | No (this computation is public) | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| User-defined keywords | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Estimated Impact | 182 | ||||||||||||||||||||||||||||||||||||||||||||||||||||
Tree of Dependent Computations | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Family? (F = Feedback message, R = changed R code, M = changed R Module, P = changed Parameters, D = changed Data) | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| F [Univariate Explorative Data Analysis] [Investigating dis...] [2007-10-22 19:45:25] [b9964c45117f7aac638ab9056d451faa] F R D [Univariate Explorative Data Analysis] [Q7] [2008-10-26 14:53:24] [6912578025c824de531bc660dd61b996] [Current] F D [Univariate Explorative Data Analysis] [Q7] [2008-10-27 19:54:21] [b47fceb71c9525e79a89b5fc6d023d0e] - PD [Univariate Explorative Data Analysis] [Blog met lags] [2008-11-01 20:54:42] [ed2ba3b6182103c15c0ab511ae4e6284] - PD [Univariate Explorative Data Analysis] [Q7] [2008-11-03 18:43:02] [b47fceb71c9525e79a89b5fc6d023d0e] F D [Univariate Explorative Data Analysis] [] [2008-10-27 20:23:41] [fd59abe368d8219a006d49608e51987e] - P [Univariate Explorative Data Analysis] [Q7 Correctie] [2008-10-30 21:15:11] [547636b63517c1c2916a747d66b36ebf] - R [Univariate Explorative Data Analysis] [Q7 waarde - gemid...] [2008-10-30 21:56:10] [547636b63517c1c2916a747d66b36ebf] - PD [Univariate Explorative Data Analysis] [] [2008-10-31 09:46:15] [a4ee3bef49b119f4bd2e925060c84f5e] - PD [Univariate Explorative Data Analysis] [tijdreeks autocor...] [2008-11-03 23:48:42] [82d201ca7b4e7cd2c6f885d29b5b6937] - RMPD [Central Tendency] [central tendency] [2008-11-04 00:10:54] [82d201ca7b4e7cd2c6f885d29b5b6937] | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Feedback Forum | |||||||||||||||||||||||||||||||||||||||||||||||||||||
Post a new message | |||||||||||||||||||||||||||||||||||||||||||||||||||||
Dataset | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Dataseries X: | |||||||||||||||||||||||||||||||||||||||||||||||||||||
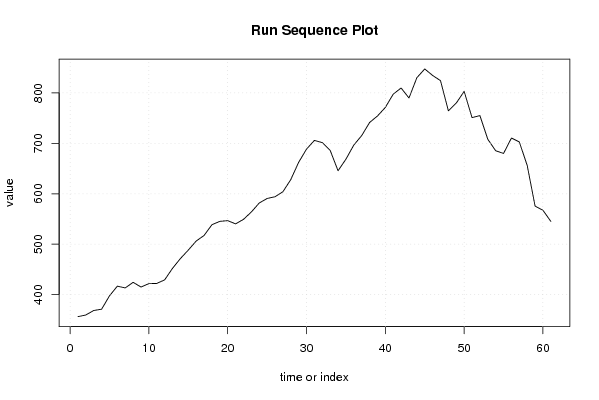
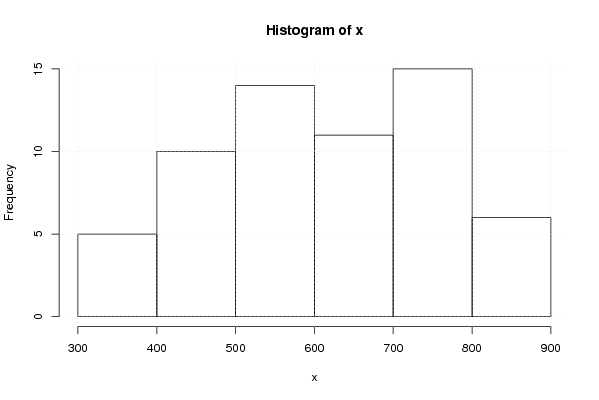
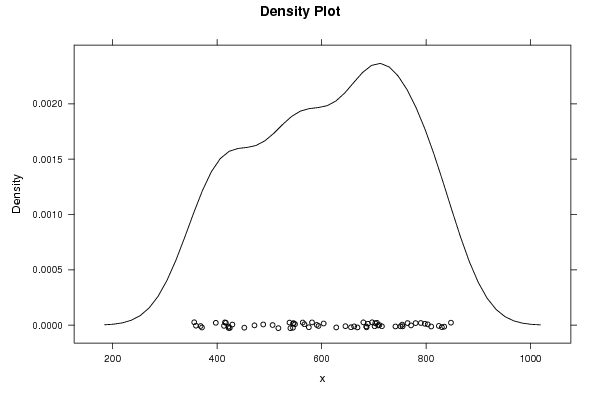
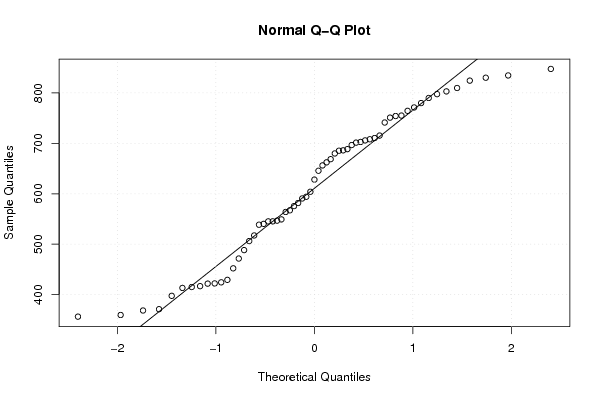
356,2 359,5 368,4 371 397,5 416,7 413,2 424,3 415 421,7 422,1 429,2 452,1 471,5 488,3 506,2 517,3 538,6 545,3 546,7 540,3 549,2 563,9 581,7 590,7 594,1 604 628,1 662,4 688,6 705,9 701,5 686,2 645,7 668,7 696,7 715,5 741,4 754,3 771,3 797,7 809,9 790,1 830,3 847,7 834,8 824,5 764,6 780 803,2 751,1 755,2 708,2 685,4 680 710,6 702,8 656,3 575,6 567,2 545,2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||
Tables (Output of Computation) | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||||
Figures (Output of Computation) | |||||||||||||||||||||||||||||||||||||||||||||||||||||
Input Parameters & R Code | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Parameters (Session): | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| par1 = 0 ; par2 = 0 ; | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Parameters (R input): | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| par1 = 0 ; par2 = 0 ; | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| R code (references can be found in the software module): | |||||||||||||||||||||||||||||||||||||||||||||||||||||
par1 <- as.numeric(par1) | |||||||||||||||||||||||||||||||||||||||||||||||||||||