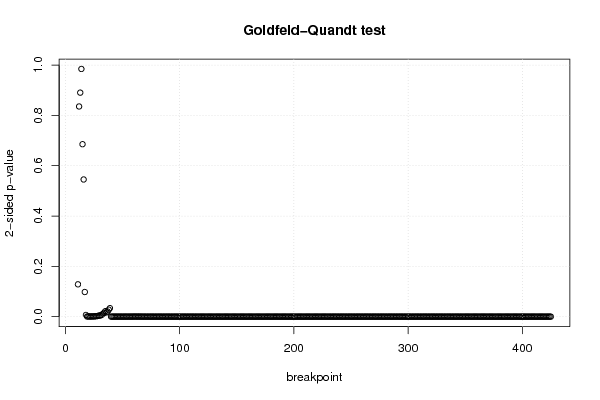
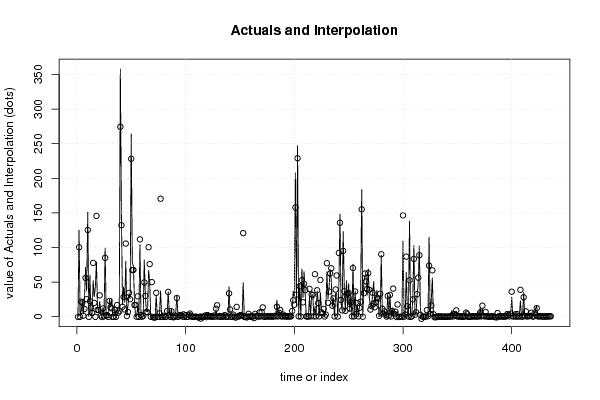
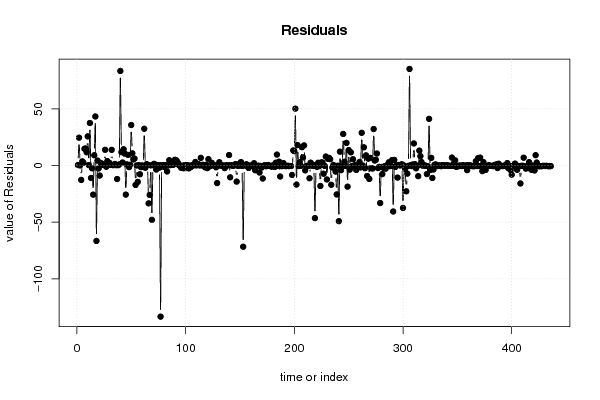
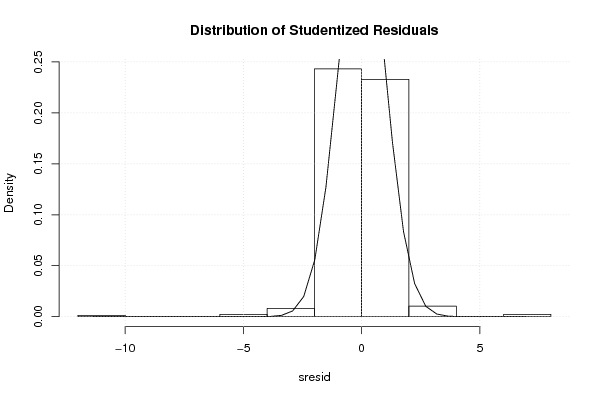
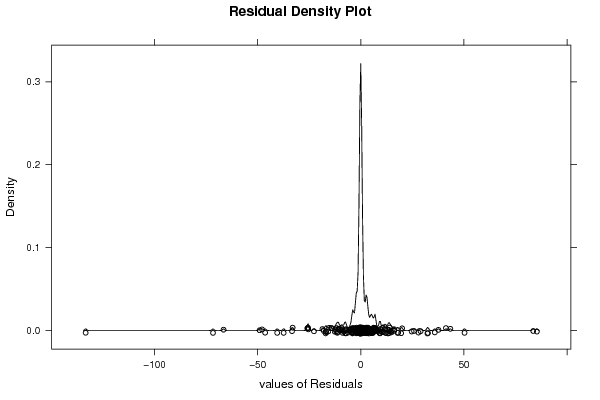
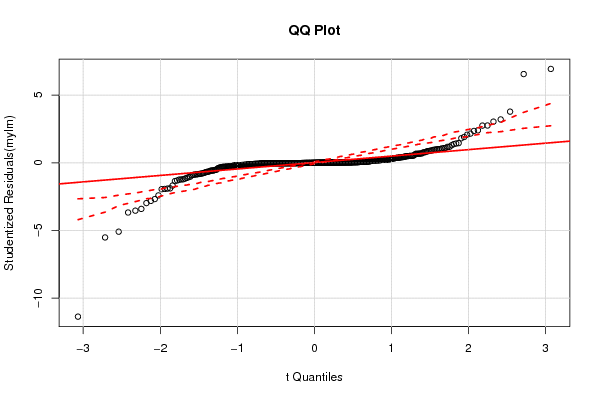
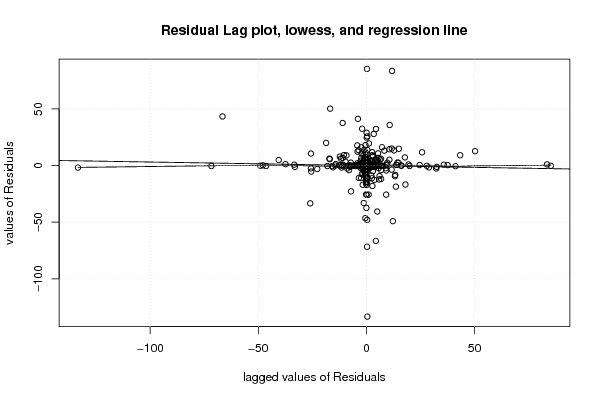
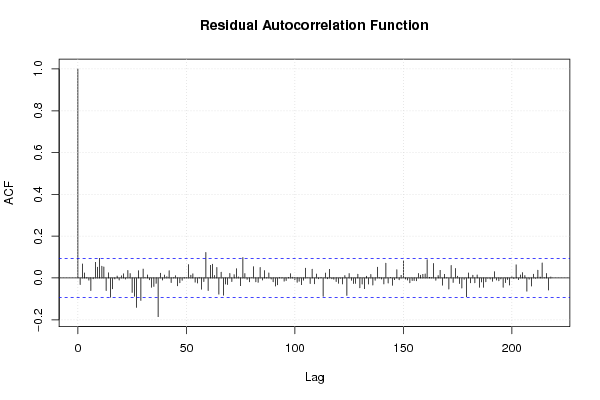
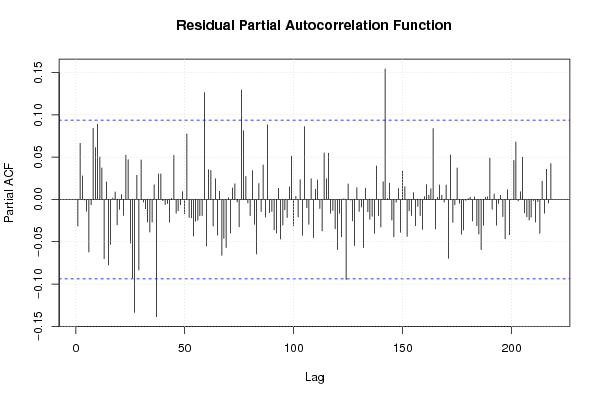
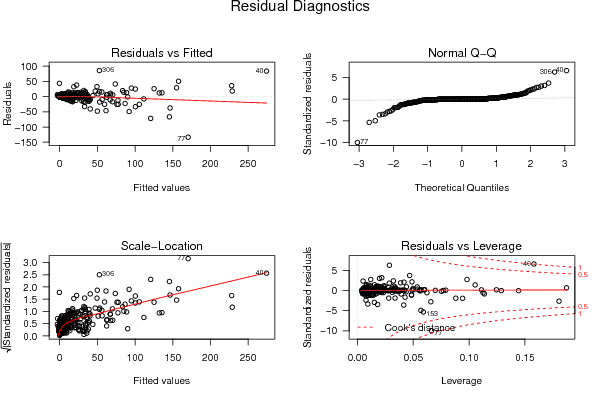
0,00 0,00 0,00 0,00 180,00 9,00 9,00 21,00
125,00 121,00 11,00 0,00 326,00 16,00 11,00 21,00
0,00 0,00 1,00 0,00 332,00 18,00 12,00 21,00
9,00 28,00 0,00 0,00 274,00 18,00 14,00 21,00
24,00 26,00 1,00 0,00 459,00 20,00 18,00 21,00
4,00 1,00 3,00 2,00 263,00 13,00 12,00 21,00
26,00 12,00 5,00 2,00 243,00 11,00 9,00 21,00
71,00 66,00 11,00 12,00 365,00 25,00 20,00 21,00
37,00 30,00 7,00 16,00 300,00 16,00 16,00 21,00
151,00 155,00 8,00 9,00 262,00 20,00 11,00 21,00
0,00 0,00 0,00 0,00 100,00 7,00 3,00 21,00
60,00 27,00 3,00 0,00 132,00 8,00 8,00 21,00
2,00 2,00 25,00 2,00 206,00 22,00 20,00 21,00
3,00 0,00 12,00 0,00 130,00 21,00 15,00 21,00
52,00 99,00 4,00 17,00 864,00 27,00 27,00 21,00
29,00 25,00 2,00 0,00 526,00 21,00 15,00 21,00
43,00 0,00 2,00 0,00 852,00 25,00 19,00 21,00
79,00 180,00 10,00 5,00 827,00 20,00 24,00 21,00
14,00 13,00 0,00 0,00 431,00 43,00 13,00 21,00
7,00 12,00 2,00 3,00 350,00 13,00 13,00 21,00
22,00 40,00 0,00 4,00 294,00 17,00 17,00 21,00
4,00 2,00 7,00 34,00 187,00 10,00 9,00 21,00
0,00 0,00 0,00 0,00 314,00 18,00 16,00 21,00
13,00 15,00 2,00 7,00 82,00 7,00 5,00 21,00
1,00 0,00 3,00 2,00 440,00 12,00 9,00 21,00
99,00 105,00 6,00 0,00 516,00 15,00 14,00 21,00
1,00 3,00 0,00 0,00 90,00 8,00 7,00 21,00
6,00 2,00 2,00 0,00 194,00 6,00 6,00 21,00
0,00 0,00 0,00 0,00 183,00 9,00 9,00 20,00
25,00 24,00 11,00 12,00 500,00 28,00 28,00 20,00
14,00 17,00 2,00 6,00 263,00 10,00 9,00 20,00
26,00 12,00 8,00 8,00 261,00 10,00 9,00 20,00
0,00 0,00 0,00 0,00 228,00 13,00 12,00 20,00
0,00 0,00 0,00 0,00 187,00 11,00 9,00 20,00
11,00 13,00 0,00 3,00 310,00 15,00 15,00 20,00
0,00 0,00 0,00 0,00 167,00 35,00 6,00 20,00
5,00 22,00 1,00 6,00 282,00 12,00 12,00 20,00
6,00 8,00 0,00 1,00 305,00 11,00 9,00 20,00
9,00 4,00 13,00 7,00 490,00 21,00 12,00 20,00
358,00 342,00 14,00 28,00 448,00 19,00 19,00 20,00
144,00 164,00 8,00 7,00 747,00 45,00 29,00 20,00
17,00 19,00 0,00 1,00 441,00 23,00 19,00 20,00
43,00 39,00 1,00 27,00 387,00 18,00 18,00 20,00
21,00 12,00 4,00 5,00 366,00 16,00 15,00 20,00
80,00 125,00 24,00 47,00 660,00 27,00 25,00 20,00
2,00 0,00 4,00 2,00 510,00 18,00 16,00 20,00
16,00 8,00 2,00 2,00 255,00 11,00 9,00 20,00
33,00 41,00 7,00 11,00 286,00 16,00 11,00 20,00
26,00 33,00 0,00 2,00 425,00 20,00 12,00 20,00
264,00 279,00 19,00 15,00 296,00 20,00 16,00 20,00
78,00 77,00 15,00 2,00 470,00 24,00 19,00 20,00
73,00 84,00 4,00 6,00 134,00 10,00 9,00 20,00
23,00 22,00 0,00 2,00 195,00 9,00 9,00 20,00
0,00 22,00 0,00 0,00 123,00 9,00 9,00 20,00
0,00 0,00 0,00 0,00 115,00 30,00 18,00 20,00
15,00 30,00 17,00 21,00 507,00 30,00 19,00 20,00
0,00 0,00 0,00 0,00 109,00 7,00 6,00 20,00
104,00 129,00 24,00 12,00 618,00 31,00 23,00 20,00
1,00 0,00 6,00 0,00 274,00 15,00 12,00 20,00
0,00 0,00 0,00 0,00 168,00 24,00 24,00 20,00
0,00 0,00 3,00 0,00 190,00 23,00 17,00 20,00
82,00 54,00 15,00 2,00 126,00 17,00 15,00 20,00
28,00 38,00 1,00 1,00 294,00 15,00 13,00 20,00
8,00 9,00 1,00 2,00 399,00 25,00 14,00 20,00
7,00 7,00 3,00 2,00 317,00 12,00 12,00 20,00
67,00 131,00 7,00 75,00 331,00 15,00 12,00 20,00
50,00 95,00 3,00 3,00 308,00 22,00 21,00 20,00
0,00 0,00 3,00 0,00 1144,00 29,00 28,00 20,00
2,00 63,00 2,00 1,00 452,00 17,00 9,00 20,00
0,00 0,00 0,00 0,00 322,00 25,00 24,00 20,00
0,00 0,00 0,00 0,00 990,00 40,00 14,00 20,00
0,00 0,00 0,00 0,00 547,00 15,00 11,00 20,00
31,00 43,00 3,00 2,00 464,00 21,00 10,00 20,00
0,00 0,00 0,00 0,00 143,00 6,00 6,00 20,00
0,00 0,00 0,00 0,00 138,00 7,00 7,00 20,00
3,00 0,00 12,00 0,00 560,00 17,00 14,00 20,00
37,00 211,00 10,00 7,00 400,00 11,00 10,00 20,00
0,00 0,00 0,00 0,00 221,00 18,00 9,00 20,00
0,00 0,00 0,00 0,00 310,00 20,00 16,00 20,00
0,00 0,00 3,00 0,00 74,00 10,00 3,00 20,00
0,00 0,00 0,00 0,00 162,00 17,00 7,00 20,00
0,00 0,00 0,00 0,00 67,00 4,00 6,00 20,00
3,00 11,00 0,00 1,00 198,00 9,00 9,00 20,00
36,00 46,00 1,00 7,00 259,00 14,00 12,00 20,00
7,00 3,00 1,00 2,00 140,00 6,00 6,00 20,00
0,00 0,00 0,00 0,00 94,00 4,00 3,00 20,00
11,00 10,00 1,00 1,00 285,00 15,00 12,00 19,00
0,00 0,00 0,00 0,00 270,00 12,00 3,00 19,00
0,00 0,00 0,00 0,00 755,00 62,00 25,00 19,00
5,00 0,00 1,00 3,00 148,00 11,00 7,00 19,00
6,00 2,00 0,00 3,00 244,00 21,00 21,00 19,00
31,00 31,00 7,00 0,00 697,00 28,00 26,00 18,00
2,00 0,00 0,00 0,00 226,00 15,00 11,00 18,00
2,00 3,00 0,00 0,00 400,00 15,00 12,00 18,00
2,00 2,00 2,00 2,00 105,00 6,00 9,00 17,00
0,00 0,00 6,00 5,00 376,00 18,00 16,00 17,00
0,00 0,00 0,00 0,00 79,00 21,00 20,00 17,00
1,00 4,00 1,00 0,00 160,00 8,00 8,00 17,00
0,00 0,00 0,00 0,00 70,00 4,00 2,00 17,00
0,00 0,00 5,00 0,00 87,00 12,00 10,00 17,00
0,00 0,00 0,00 0,00 234,00 10,00 10,00 17,00
0,00 0,00 0,00 0,00 223,00 7,00 1,00 16,00
0,00 0,00 7,00 0,00 682,00 26,00 18,00 16,00
4,00 6,00 0,00 1,00 149,00 9,00 9,00 16,00
0,00 0,00 3,00 0,00 195,00 16,00 15,00 16,00
0,00 0,00 0,00 0,00 236,00 5,00 5,00 16,00
0,00 0,00 0,00 0,00 112,00 7,00 7,00 16,00
0,00 0,00 0,00 0,00 184,00 4,00 4,00 16,00
3,00 0,00 0,00 0,00 65,00 6,00 3,00 16,00
0,00 0,00 0,00 0,00 62,00 3,00 3,00 15,00
0,00 0,00 0,00 0,00 424,00 29,00 5,00 15,00
0,00 0,00 0,00 0,00 906,00 26,00 15,00 15,00
0,00 0,00 0,00 0,00 271,00 43,00 17,00 15,00
4,00 1,00 0,00 34,00 712,00 17,00 17,00 14,00
0,00 0,00 0,00 0,00 200,00 10,00 9,00 13,00
0,00 0,00 0,00 0,00 520,00 12,00 3,00 13,00
0,00 0,00 0,00 0,00 113,00 7,00 7,00 13,00
0,00 2,00 0,00 0,00 89,00 15,00 5,00 13,00
0,00 0,00 0,00 0,00 600,00 20,00 13,00 12,00
0,00 3,00 0,00 0,00 148,00 8,00 6,00 12,00
6,00 0,00 5,00 13,00 912,00 35,00 22,00 12,00
0,00 2,00 0,00 0,00 719,00 17,00 15,00 12,00
0,00 0,00 0,00 0,00 137,00 14,00 3,00 11,00
3,00 3,00 0,00 1,00 1480,00 33,00 29,00 11,00
0,00 0,00 0,00 0,00 243,00 11,00 10,00 11,00
2,00 2,00 0,00 0,00 172,00 13,00 12,00 11,00
0,00 0,00 0,00 0,00 201,00 10,00 9,00 11,00
10,00 14,00 3,00 11,00 174,00 10,00 5,00 11,00
1,00 21,00 0,00 3,00 106,00 10,00 9,00 11,00
0,00 0,00 0,00 0,00 195,00 10,00 9,00 10,00
4,00 0,00 2,00 1,00 65,00 9,00 9,00 10,00
0,00 0,00 0,00 0,00 153,00 6,00 6,00 10,00
0,00 0,00 0,00 0,00 357,00 19,00 18,00 9,00
0,00 0,00 0,00 0,00 495,00 23,00 22,00 9,00
0,00 0,00 1,00 0,00 105,00 10,00 10,00 9,00
0,00 0,00 5,00 0,00 283,00 9,00 9,00 9,00
0,00 0,00 0,00 0,00 291,00 6,00 6,00 9,00
0,00 0,00 0,00 0,00 375,00 53,00 16,00 9,00
0,00 0,00 0,00 0,00 326,00 7,00 8,00 9,00
43,00 40,00 4,00 1,00 53,00 7,00 9,00 9,00
0,00 13,00 0,00 0,00 179,00 15,00 10,00 8,00
0,00 0,00 0,00 0,00 440,00 10,00 10,00 8,00
0,00 0,00 0,00 0,00 161,00 14,00 12,00 8,00
3,00 5,00 3,00 15,00 1233,00 35,00 25,00 8,00
0,00 0,00 0,00 0,00 260,00 13,00 8,00 8,00
0,00 0,00 0,00 0,00 1211,00 22,00 19,00 8,00
0,00 18,00 0,00 0,00 389,00 16,00 19,00 8,00
0,00 0,00 0,00 0,00 158,00 13,00 12,00 8,00
0,00 0,00 0,00 0,00 78,00 8,00 4,00 8,00
4,00 0,00 4,00 0,00 102,00 9,00 9,00 8,00
5,00 0,00 4,00 1,00 165,00 10,00 9,00 8,00
1,00 1,00 1,00 1,00 74,00 4,00 4,00 7,00
49,00 140,00 21,00 1,00 252,00 11,00 9,00 7,00
0,00 0,00 0,00 0,00 453,00 12,00 9,00 7,00
0,00 0,00 0,00 0,00 270,00 9,00 8,00 7,00
0,00 0,00 0,00 0,00 1230,00 42,00 28,00 7,00
0,00 0,00 0,00 0,00 365,00 37,00 19,00 7,00
2,00 1,00 7,00 0,00 171,00 3,00 3,00 7,00
0,00 0,00 0,00 0,00 53,00 7,00 7,00 6,00
0,00 0,00 0,00 0,00 402,00 10,00 8,00 6,00
0,00 0,00 0,00 0,00 91,00 9,00 9,00 6,00
0,00 0,00 0,00 0,00 345,00 19,00 17,00 5,00
0,00 0,00 0,00 0,00 1943,00 39,00 39,00 5,00
0,00 5,00 0,00 0,00 232,00 17,00 12,00 5,00
0,00 1,00 0,00 0,00 136,00 9,00 9,00 5,00
0,00 0,00 1,00 0,00 120,00 1,00 1,00 5,00
0,00 0,00 0,00 0,00 404,00 17,00 16,00 3,00
0,00 0,00 13,00 2,00 385,00 19,00 19,00 3,00
0,00 0,00 0,00 0,00 270,00 20,00 20,00 3,00
5,00 4,00 7,00 1,00 125,00 10,00 6,00 3,00
2,00 13,00 6,00 0,00 130,00 7,00 6,00 3,00
0,00 0,00 0,00 0,00 268,00 5,00 4,00 3,00
0,00 0,00 0,00 0,00 680,00 30,00 18,00 2,00
0,00 0,00 1,00 0,00 320,00 20,00 9,00 2,00
0,00 0,00 0,00 0,00 196,00 6,00 9,00 2,00
1,00 0,00 1,00 1,00 451,00 20,00 20,00 1,00
0,00 0,00 0,00 0,00 278,00 19,00 11,00 1,00
0,00 0,00 0,00 0,00 194,00 14,00 10,00 1,00
0,00 0,00 1,00 0,00 92,00 7,00 9,00 1,00
0,00 0,00 1,00 0,00 844,00 23,00 13,00 1,00
0,00 0,00 0,00 0,00 137,00 13,00 11,00 1,00
0,00 0,00 3,00 0,00 565,00 13,00 12,00 1,00
3,00 0,00 0,00 0,00 55,00 4,00 3,00 1,00
24,00 17,00 2,00 2,00 520,00 25,00 20,00 0,00
0,00 0,00 0,00 0,00 277,00 20,00 17,00 0,00
9,00 4,00 4,00 1,00 71,00 6,00 9,00 0,00
0,00 12,00 0,00 0,00 435,00 20,00 18,00 0,00
0,00 0,00 0,00 0,00 122,00 10,00 9,00 0,00
0,00 0,00 0,00 0,00 104,00 9,00 3,00 0,00
5,00 2,00 2,00 1,00 185,00 10,00 10,00 0,00
0,00 0,00 0,00 0,00 227,00 13,00 13,00 0,00
0,00 0,00 0,00 0,00 262,00 17,00 17,00 0,00
0,00 0,00 0,00 0,00 83,00 5,00 4,00 0,00
0,00 0,00 0,00 0,00 383,00 20,00 9,00 0,00
0,00 0,00 0,00 0,00 51,00 3,00 3,00 0,00
0,00 0,00 0,00 0,00 267,00 15,00 15,00 0,00
0,00 0,00 0,00 0,00 195,00 17,00 10,00 0,00
0,00 10,00 0,00 0,00 192,00 8,00 8,00 0,00
37,00 28,00 4,00 9,00 275,00 24,00 9,00 0,00
30,00 22,00 1,00 10,00 455,00 21,00 19,00 0,00
208,00 193,00 13,00 19,00 295,00 16,00 16,00 0,00
15,00 39,00 5,00 20,00 485,00 26,00 20,00 0,00
247,00 281,00 15,00 9,00 451,00 19,00 19,00 0,00
0,00 0,00 0,00 0,00 162,00 9,00 9,00 0,00
47,00 47,00 14,00 0,00 520,00 25,00 25,00 0,00
0,00 0,00 0,00 0,00 350,00 14,00 14,00 0,00
69,00 64,00 4,00 1,00 249,00 12,00 11,00 0,00
28,00 26,00 0,00 2,00 205,00 11,00 9,00 0,00
65,00 56,00 7,00 9,00 395,00 24,00 21,00 0,00
34,00 41,00 13,00 7,00 585,00 49,00 30,00 0,00
0,00 0,00 0,00 0,00 100,00 7,00 4,00 0,00
0,00 0,00 0,00 0,00 511,00 27,00 27,00 0,00
0,00 0,00 0,00 0,00 327,00 18,00 18,00 0,00
29,00 46,00 8,00 5,00 178,00 12,00 9,00 0,00
3,00 0,00 0,00 0,00 112,00 6,00 5,00 0,00
32,00 40,00 1,00 4,00 365,00 24,00 17,00 0,00
32,00 33,00 12,00 9,00 348,00 15,00 15,00 0,00
0,00 0,00 0,00 0,00 198,00 28,00 6,00 0,00
15,00 73,00 8,00 6,00 265,00 14,00 12,00 0,00
0,00 0,00 0,00 0,00 57,00 3,00 3,00 0,00
37,00 44,00 6,00 0,00 110,00 9,00 6,00 0,00
22,00 21,00 7,00 10,00 290,00 17,00 17,00 0,00
0,00 0,00 0,00 0,00 145,00 7,00 7,00 0,00
35,00 64,00 5,00 3,00 368,00 17,00 17,00 0,00
9,00 6,00 4,00 9,00 295,00 15,00 14,00 0,00
7,00 5,00 2,00 7,00 522,00 23,00 20,00 0,00
4,00 7,00 11,00 0,00 137,00 7,00 8,00 0,00
0,00 0,00 0,00 3,00 71,00 7,00 9,00 0,00
11,00 3,00 1,00 1,00 340,00 16,00 16,00 0,00
65,00 92,00 9,00 3,00 143,00 7,00 9,00 0,00
26,00 23,00 3,00 0,00 58,00 4,00 3,00 0,00
43,00 36,00 16,00 2,00 327,00 20,00 17,00 0,00
68,00 70,00 15,00 10,00 107,00 21,00 19,00 0,00
53,00 84,00 7,00 4,00 150,00 13,00 11,00 0,00
14,00 17,00 4,00 0,00 241,00 15,00 12,00 0,00
27,00 29,00 7,00 1,00 399,00 30,00 18,00 0,00
0,00 0,00 0,00 0,00 147,00 8,00 6,00 0,00
34,00 42,00 13,00 5,00 382,00 27,00 19,00 0,00
34,00 72,00 6,00 4,00 895,00 67,00 47,00 0,00
0,00 0,00 0,00 0,00 430,00 17,00 17,00 0,00
43,00 118,00 0,00 20,00 340,00 21,00 19,00 0,00
148,00 167,00 7,00 2,00 200,00 11,00 10,00 0,00
20,00 28,00 8,00 27,00 440,00 32,00 28,00 0,00
9,00 11,00 0,00 0,00 111,00 12,00 9,00 0,00
123,00 117,00 6,00 5,00 705,00 43,00 33,00 0,00
40,00 39,00 14,00 14,00 413,00 23,00 19,00 0,00
8,00 10,00 0,00 0,00 171,00 12,00 10,00 0,00
52,00 40,00 0,00 0,00 204,00 10,00 8,00 0,00
15,00 41,00 2,00 3,00 143,00 15,00 9,00 0,00
48,00 44,00 1,00 15,00 430,00 23,00 23,00 0,00
8,00 14,00 2,00 10,00 193,00 11,00 9,00 0,00
32,00 17,00 15,00 7,00 284,00 14,00 13,00 0,00
0,00 0,00 0,00 0,00 242,00 15,00 14,00 0,00
76,00 86,00 4,00 1,00 106,00 8,00 9,00 0,00
0,00 0,00 0,00 0,00 174,00 12,00 10,00 0,00
37,00 43,00 4,00 1,00 68,00 5,00 9,00 0,00
0,00 4,00 1,00 0,00 384,00 19,00 17,00 0,00
18,00 24,00 1,00 2,00 322,00 14,00 14,00 0,00
0,00 0,00 0,00 0,00 70,00 3,00 1,00 0,00
16,00 14,00 3,00 1,00 210,00 14,00 11,00 0,00
20,00 31,00 0,00 39,00 425,00 24,00 21,00 0,00
184,00 191,00 11,00 20,00 282,00 12,00 12,00 0,00
0,00 0,00 0,00 0,00 692,00 27,00 27,00 0,00
50,00 37,00 12,00 17,00 430,00 31,00 23,00 0,00
60,00 76,00 8,00 23,00 396,00 22,00 18,00 0,00
60,00 61,00 6,00 5,00 382,00 19,00 19,00 0,00
31,00 48,00 5,00 6,00 238,00 14,00 12,00 0,00
69,00 72,00 14,00 14,00 305,00 22,00 21,00 0,00
27,00 48,00 7,00 33,00 329,00 15,00 8,00 0,00
17,00 12,00 2,00 10,00 75,00 7,00 9,00 0,00
13,00 16,00 6,00 7,00 174,00 11,00 9,00 0,00
32,00 40,00 6,00 3,00 311,00 23,00 14,00 0,00
51,00 22,00 3,00 6,00 209,00 14,00 9,00 0,00
18,00 17,00 0,00 4,00 128,00 6,00 6,00 0,00
26,00 24,00 4,00 2,00 479,00 19,00 19,00 0,00
38,00 34,00 5,00 27,00 355,00 16,00 16,00 0,00
24,00 34,00 0,00 10,00 489,00 27,00 18,00 0,00
0,00 0,00 2,00 1,00 310,00 17,00 13,00 0,00
0,00 40,00 3,00 1,00 473,00 18,00 18,00 0,00
89,00 105,00 14,00 1,00 145,00 18,00 14,00 0,00
4,00 2,00 21,00 8,00 206,00 47,00 19,00 0,00
2,00 0,00 8,00 3,00 140,00 16,00 13,00 0,00
7,00 9,00 0,00 0,00 130,00 8,00 8,00 0,00
0,00 3,00 1,00 7,00 510,00 55,00 39,00 0,00
0,00 0,00 0,00 0,00 500,00 25,00 18,00 0,00
31,00 35,00 5,00 3,00 451,00 18,00 18,00 0,00
4,00 1,00 0,00 2,00 96,00 5,00 5,00 0,00
31,00 34,00 9,00 0,00 208,00 8,00 8,00 0,00
0,00 0,00 0,00 0,00 251,00 16,00 4,00 0,00
13,00 9,00 2,00 5,00 156,00 9,00 9,00 0,00
0,00 51,00 0,00 0,00 428,00 22,00 20,00 0,00
9,00 3,00 3,00 3,00 140,00 8,00 7,00 0,00
6,00 5,00 7,00 6,00 300,00 23,00 20,00 0,00
0,00 0,00 0,00 0,00 838,00 19,00 19,00 0,00
7,00 20,00 3,00 0,00 208,00 11,00 10,00 0,00
0,00 0,00 0,00 0,00 897,00 21,00 21,00 0,00
0,00 0,00 0,00 0,00 674,00 25,00 18,00 0,00
0,00 0,00 0,00 0,00 622,00 20,00 14,00 0,00
1,00 0,00 1,00 2,00 877,00 24,00 17,00 0,00
109,00 183,00 5,00 3,00 873,00 19,00 19,00 0,00
0,00 0,00 0,00 0,00 461,00 13,00 11,00 0,00
0,00 0,00 6,00 0,00 302,00 9,00 9,00 0,00
64,00 102,00 14,00 0,00 997,00 28,00 24,00 0,00
8,00 19,00 2,00 3,00 980,00 32,00 21,00 0,00
0,00 0,00 0,00 0,00 235,00 3,00 3,00 0,00
138,00 62,00 9,00 1,00 917,00 19,00 19,00 0,00
0,00 0,00 0,00 0,00 673,00 21,00 14,00 0,00
0,00 0,00 0,00 0,00 100,00 9,00 9,00 0,00
26,00 30,00 5,00 18,00 203,00 10,00 9,00 0,00
103,00 100,00 9,00 3,00 310,00 17,00 14,00 0,00
4,00 2,00 2,00 0,00 267,00 11,00 11,00 0,00
4,00 8,00 0,00 3,00 177,00 7,00 7,00 0,00
32,00 45,00 4,00 64,00 429,00 17,00 17,00 0,00
47,00 68,00 5,00 2,00 227,00 10,00 9,00 0,00
102,00 106,00 10,00 1,00 365,00 17,00 17,00 0,00
11,00 8,00 5,00 75,00 565,00 26,00 21,00 0,00
0,00 0,00 1,00 38,00 898,00 26,00 23,00 0,00
0,00 0,00 0,00 0,00 410,00 16,00 11,00 0,00
0,00 0,00 0,00 0,00 665,00 17,00 11,00 0,00
0,00 0,00 0,00 0,00 543,00 17,00 15,00 0,00
0,00 0,00 0,00 0,00 312,00 9,00 9,00 0,00
2,00 11,00 1,00 1,00 129,00 5,00 6,00 0,00
0,00 0,00 0,00 0,00 127,00 23,00 13,00 0,00
115,00 90,00 6,00 2,00 538,00 10,00 10,00 0,00
0,00 5,00 0,00 0,00 529,00 17,00 16,00 0,00
23,00 21,00 1,00 9,00 605,00 16,00 16,00 0,00
56,00 81,00 7,00 6,00 442,00 13,00 10,00 0,00
0,00 2,00 3,00 0,00 132,00 9,00 9,00 0,00
0,00 0,00 0,00 0,00 986,00 25,00 23,00 0,00
0,00 0,00 0,00 0,00 1060,00 19,00 8,00 0,00
0,00 0,00 0,00 0,00 765,00 13,00 15,00 0,00
0,00 0,00 0,00 0,00 140,00 10,00 7,00 0,00
0,00 0,00 0,00 0,00 558,00 17,00 17,00 0,00
0,00 0,00 0,00 0,00 480,00 11,00 10,00 0,00
0,00 0,00 0,00 0,00 78,00 10,00 9,00 0,00
0,00 0,00 0,00 0,00 572,00 13,00 13,00 0,00
0,00 0,00 0,00 0,00 667,00 22,00 11,00 0,00
0,00 0,00 0,00 0,00 542,00 13,00 13,00 0,00
0,00 0,00 0,00 0,00 236,00 4,00 3,00 0,00
0,00 0,00 0,00 0,00 548,00 20,00 6,00 0,00
0,00 0,00 0,00 0,00 618,00 17,00 16,00 0,00
0,00 0,00 0,00 0,00 254,00 5,00 3,00 0,00
0,00 0,00 0,00 0,00 235,00 12,00 6,00 0,00
0,00 0,00 0,00 0,00 329,00 11,00 9,00 0,00
9,00 5,00 0,00 28,00 217,00 5,00 5,00 0,00
0,00 0,00 0,00 0,00 276,00 9,00 9,00 0,00
6,00 5,00 0,00 3,00 486,00 11,00 11,00 0,00
6,00 0,00 2,00 0,00 87,00 5,00 5,00 0,00
8,00 10,00 3,00 6,00 238,00 10,00 9,00 0,00
0,00 0,00 0,00 0,00 228,00 15,00 11,00 0,00
0,00 0,00 1,00 1,00 385,00 18,00 17,00 0,00
0,00 0,00 0,00 0,00 620,00 22,00 22,00 0,00
0,00 0,00 0,00 0,00 143,00 8,00 2,00 0,00
0,00 0,00 0,00 0,00 250,00 19,00 18,00 0,00
0,00 0,00 0,00 0,00 1045,00 48,00 44,00 0,00
0,00 0,00 0,00 0,00 322,00 8,00 8,00 0,00
0,00 0,00 0,00 0,00 80,00 3,00 2,00 0,00
6,00 6,00 3,00 6,00 334,00 9,00 9,00 0,00
0,00 0,00 9,00 0,00 627,00 18,00 17,00 0,00
0,00 0,00 0,00 0,00 729,00 17,00 17,00 0,00
0,00 0,00 0,00 0,00 430,00 9,00 9,00 0,00
0,00 0,00 0,00 0,00 519,00 16,00 12,00 0,00
0,00 0,00 0,00 0,00 212,00 5,00 5,00 0,00
0,00 0,00 0,00 0,00 72,00 10,00 9,00 0,00
0,00 0,00 0,00 0,00 133,00 13,00 15,00 0,00
0,00 0,00 0,00 0,00 135,00 8,00 7,00 0,00
4,00 0,00 0,00 3,00 112,00 8,00 4,00 0,00
0,00 0,00 0,00 0,00 84,00 10,00 1,00 0,00
7,00 0,00 0,00 0,00 81,00 8,00 7,00 0,00
0,00 0,00 0,00 0,00 165,00 9,00 9,00 0,00
13,00 7,00 0,00 0,00 138,00 9,00 9,00 0,00
0,00 0,00 0,00 0,00 163,00 9,00 9,00 0,00
11,00 18,00 3,00 1,00 285,00 15,00 13,00 0,00
4,00 1,00 0,00 3,00 113,00 5,00 6,00 0,00
0,00 0,00 0,00 0,00 203,00 14,00 9,00 0,00
3,00 6,00 4,00 3,00 90,00 7,00 5,00 0,00
0,00 0,00 0,00 0,00 121,00 7,00 1,00 0,00
0,00 0,00 0,00 0,00 297,00 18,00 17,00 0,00
0,00 0,00 0,00 0,00 760,00 17,00 16,00 0,00
0,00 0,00 0,00 0,00 266,00 9,00 8,00 0,00
0,00 0,00 0,00 0,00 219,00 9,00 9,00 0,00
0,00 0,00 0,00 0,00 111,00 1,00 1,00 0,00
0,00 0,00 0,00 0,00 69,00 5,00 9,00 0,00
0,00 0,00 0,00 0,00 840,00 19,00 17,00 0,00
0,00 0,00 0,00 0,00 755,00 14,00 16,00 0,00
0,00 0,00 0,00 0,00 1362,00 40,00 26,00 0,00
3,00 6,00 0,00 0,00 102,00 8,00 3,00 0,00
2,00 0,00 0,00 0,00 221,00 15,00 12,00 0,00
0,00 0,00 0,00 0,00 110,00 7,00 10,00 0,00
0,00 0,00 0,00 1,00 244,00 9,00 6,00 0,00
0,00 0,00 0,00 0,00 70,00 3,00 3,00 0,00
0,00 0,00 0,00 0,00 102,00 5,00 5,00 0,00
0,00 0,00 0,00 0,00 398,00 9,00 5,00 0,00
0,00 0,00 0,00 0,00 91,00 3,00 3,00 0,00
0,00 0,00 0,00 0,00 66,00 9,00 1,00 0,00
6,00 3,00 2,00 1,00 223,00 60,00 15,00 0,00
0,00 0,00 4,00 1,00 88,00 6,00 6,00 0,00
0,00 0,00 0,00 0,00 133,00 8,00 7,00 0,00
0,00 1,00 6,00 2,00 82,00 7,00 9,00 0,00
28,00 44,00 2,00 1,00 261,00 15,00 12,00 0,00
0,00 0,00 0,00 0,00 214,00 12,00 9,00 0,00
0,00 0,00 0,00 0,00 51,00 9,00 3,00 0,00
5,00 4,00 0,00 5,00 84,00 7,00 6,00 0,00
0,00 0,00 0,00 0,00 557,00 34,00 16,00 0,00
0,00 0,00 7,00 1,00 66,00 3,00 4,00 0,00
0,00 0,00 0,00 0,00 208,00 8,00 7,00 0,00
0,00 0,00 0,00 0,00 112,00 3,00 3,00 0,00
23,00 48,00 7,00 37,00 97,00 9,00 5,00 0,00
0,00 0,00 0,00 0,00 266,00 6,00 5,00 0,00
0,00 0,00 0,00 0,00 296,00 11,00 9,00 0,00
35,00 32,00 5,00 2,00 65,00 5,00 5,00 0,00
0,00 0,00 0,00 0,00 215,00 67,00 20,00 0,00
5,00 9,00 2,00 3,00 537,00 11,00 11,00 0,00
0,00 0,00 0,00 0,00 55,00 3,00 6,00 0,00
0,00 0,00 0,00 0,00 182,00 4,00 4,00 0,00
4,00 1,00 0,00 2,00 190,00 7,00 7,00 0,00
2,00 0,00 2,00 1,00 340,00 16,00 14,00 0,00
2,00 4,00 5,00 2,00 206,00 6,00 7,00 0,00
0,00 0,00 0,00 0,00 188,00 7,00 8,00 0,00
0,00 0,00 0,00 0,00 360,00 12,00 12,00 0,00
0,00 0,00 9,00 0,00 408,00 12,00 10,00 0,00
12,00 2,00 2,00 2,00 104,00 6,00 6,00 0,00
15,00 16,00 2,00 20,00 225,00 10,00 10,00 0,00
0,00 0,00 0,00 0,00 73,00 3,00 3,00 0,00
0,00 0,00 0,00 0,00 51,00 4,00 6,00 0,00
0,00 0,00 0,00 0,00 66,00 9,00 7,00 0,00
0,00 0,00 0,00 0,00 95,00 10,00 7,00 0,00
0,00 0,00 0,00 0,00 97,00 5,00 5,00 0,00
0,00 0,00 0,00 0,00 280,00 6,00 6,00 0,00
0,00 0,00 0,00 0,00 142,00 5,00 3,00 0,00
0,00 0,00 0,00 0,00 130,00 6,00 6,00 0,00
0,00 0,00 0,00 0,00 337,00 9,00 9,00 0,00
0,00 0,00 0,00 0,00 54,00 7,00 3,00 0,00
0,00 0,00 0,00 0,00 73,00 6,00 7,00 0,00
0,00 0,00 0,00 0,00 65,00 7,00 7,00 0,00
0,00 0,00 0,00 0,00 60,00 3,00 3,00 0,00
|